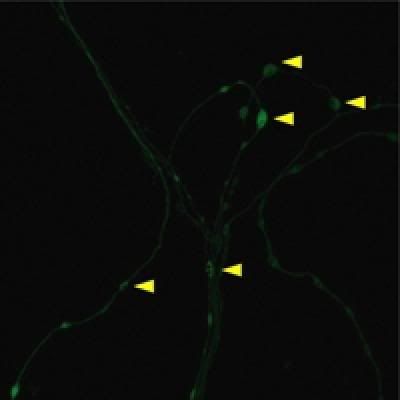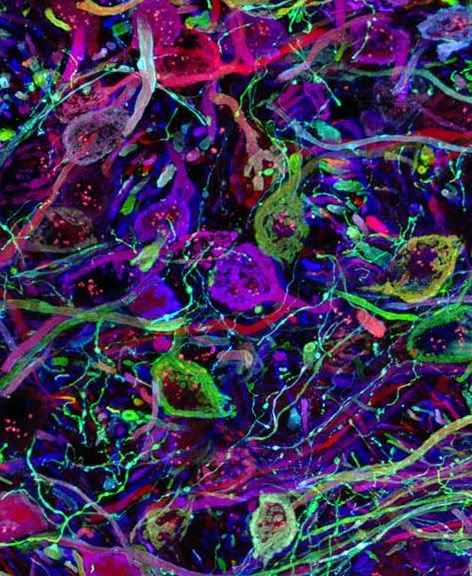
Las neuronas en los cerebros jóvenes forman una marabunta de interconexiones lanzadas en todas direcciones, con múltiples células nerviosas convergiendo en una sola célula diana. Conforme los cerebros maduran, algunas de estas conexiones se podan como las ramas inútiles de un árbol. El poder visualizar este proceso de poda en células individuales podría ayudar a los investigadores a dar una respuesta a cuestiones fundamentales acerca de cómo se propaga la información en una red neuronal. Sin embargo, llegar a conocer cómo las diferentes conexiones se ven reforzadas o eliminadas es algo bastante complicado, básicamente porque, cuando se usa una tinción para una neurona y su vecina, lo que se ve en el microscopio es un borrón indiscernible.
Y entonces va el equipo de Jeff Lichtman en 2007 y presenta el Brainbow [1]. Lichtman, un neurobiólogo de
Lo que sigue es predecible. La activación de la expresión de cre hace que la proteína corte diferentes partes del ADN-Brainbow y que exprese una proteína o roja, o azul o verde. El equipo, en el trabajo original publicado en Nature, colocó entre 8 y 16 copias de estos genes Brainbow en ratones. Lichtman comentó el efecto en términos pictóricos: “El número de copias es como pinceladas de pintura; el número de gotas de un color determina el color final expresado por la neurona”. Con múltiples copias de segmentos de ADN, el cerebro del ratón se puede pintar con 100 matices de color, lo que permite a los investigadores, usando un microscopio óptico, distinguir entre neuronas vecinas.
“¡Esto es la revolución! ¡La releche! ¡Que le den el Nobel!” eran expresiones habituales entre los que hacíamos el master en neurociencias por aquella época. La gente de prestigio también comentaba. Olaf Sporns de
Desde que se publicó Brainbow, otros equipos han comenzado a usarlo para codificar células con colores en otros organismos modelo, incluyendo el pez cebra (Danio rerio) y
Alex Schier (neurobiólogo, Harvard) ha estado usando la técnica con el pez cebra. Como el pez es transparente se puede observar el juego de colores a medida que el espécimen se desarrolla. Uno de los atractivos de la técnica Brainbow es el potencial para trazar las conexiones a larga distancia entre neuronas. Es como si intentásemos seguir con la vista un espagueti concreto en un plato lleno, sería imposible; pero si cada uno es de un color es mucho más fácil. Ése es el uso que le da Schier a la técnica para estudiar el desarrollo del sistema nervioso del pez cebra.
Pero hay grupos que son escépticos sobre el verdadero alcance de la técnica. Uno de los líderes del movimiento escéptico es Partha Mitra de los Laboratorios Cold Spring Harbour (Nueva York, EE.UU.). Su principal crítica es que no hay colores suficientes. 100 no bastan cuando se quieren estudiar entramados de miles y miles de neuronas presentes en el corte más fino de sustancia cerebral. Dado que más de una conexión neuronal podría estar pintada con el mismo tono de rojo, por ejemplo, los algoritmos computacionales tendrían que empezar a tomar riesgos: adivinar qué encaja con qué. La puerta que se abre al error es enorme.
En marzo de este año, Mitra y otros 37 científicos publicaron un artículo en PLoS Computational Biology haciendo una llamada para realizar un esfuerzo sistemático para trazar los circuitos neurales del ratón [2]. En vez de usar métodos en tecnicolor como Brainbow, el grupo pide que se desarrollen sistemas de alto rendimiento para el tratamiento de imágenes tradicionales, en las que una tintura trazadora de un solo color se inyecta en el sistema nervioso. La técnica está más establecida y es menos laboriosa que
Como dijo Svoboda (biofísico, Howard Hugues - Janelia Farm) y coautor del artículo de Mitra: “ahora mismo no es más que un montón de fotos preciosas, pero es muy prometedor”. Sí, sólo tiene que encontrar su nicho...
Referencias:
[1]
Livet, J., Weissman, T., Kang, H., Draft, R., Lu, J., Bennis, R., Sanes, J., & Lichtman, J. (2007). Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system Nature, 450 (7166), 56-62 DOI: 10.1038/nature06293